아지즈 산자르 목차 연구업적 참조 둘러보기 메뉴zinc fingerhelix turn helixmotifhelix turn helixhelicasezinc fingerhelix turn helixleucine zipperDNA-dependent ATPasehelicaseligase아지즈 산자르eheh
1946년 태어남살아있는 사람터키의 생화학자터키의 의학자터키의 무슬림터키의 노벨상 수상자노벨 화학상 수상자이스탄불 대학교 동문텍사스 대학교 동문터키계 미국인미국 과학 아카데미의 회원미국 예술과학 아카데미 석학회원이스탄불 공과대학교 동문
터키생화학자노벨 화학상Prokaryoteeukaryote아지즈 산자르zinc fingerhelix turn helixmotifhelix turn helixhelicase뉴클레오티드helicaseRNA 중합효소RNA 중합효소전사RNA 중합효소RNA 중합효소RNA 중합효소RNA 중합효소
아지즈 산자르
둘러보기로 가기
검색하러 가기
아지즈 산자르(터키어: Aziz Sancar, 1946년 9월 8일 ~ )는 터키의 생화학자다. 2015년 Nucleotide excision repair를 연구한 공로로 노벨 화학상을 수상하였다. 현재 노스캐롤라이나 대학교 생화학과 교수로 재직 중이다.
목차
1 연구업적
1.1 손상부위의 인식(Damage Recognition)
1.2 절제 수선(Excision Repair)
1.2.1 대장균에서의 절제 수선(Excision Repair in E.coli)
1.2.2 인간에서의 절제 수선(Excision Repair in Humans)
1.3 Transcription coupled repair
1.3.1 대장균의 경우(in E.coli)
1.3.2 사람의 경우(in Humans)
2 참조
연구업적
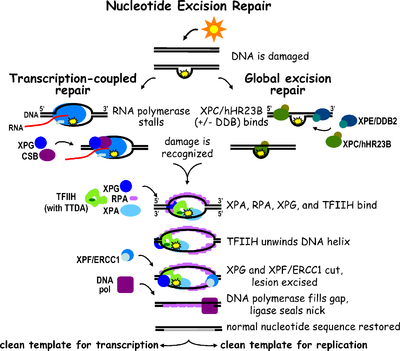
인간에게서의 절제 수선(Excision Repair in Humans)두 가지 경로로 일어난다. 과정은 # XPA, RPA XPC-TFIIH가 랜덤한 순서로 목표 지역(lesion site)에 집합한다. # XPA, RPA, XPC의 협동적 상호작용(cooperative interaction)과 TFHII의 kinetic proofreading activity(오류를 찾는 과정)에 의해 손상 부위(damage site)에 대한 특이성(specificity)를 형성한다. # 그 다음 TFIIH가 ATP 분해로 DNA를 unwinding하여 bubble을 형성하는데 이 때의 complex를 PIC I이라 칭한다. # 이 후 XPC가 XPG로 교체되어 PIC3 complex를 형성한다. 이 때 절개가 일어나 24-32nt의 oligomer가 나오고 생긴 gap은 RFC/PCNA, Pol delta-epsilon에 의해 매꿔진다. 마지막으로 DNA ligase가 봉합한다. 를 통해 일어난다.
Nucleotide excision repair(NER)는 손상이 생긴 DNA를 수선하는 기작이다. DNA는 RNA로 전사된 후 단백질을 발현하게 되고, 이 단백질이 생명체에게 필요한 여러 생리작용을 일으킨다. 때문에 DNA 손상은 개체에 매우 치명적이며, 이 손상을 계속해서 수리하는 기작이 필요하다. 생명이 탄생한 이후로 이 과정은 계속하여 진화해 왔다. Prokaryote와 eukaryote는 진화적으로 같지 않지만, 기본적으로 같은 원리의 기작을 가진다. 순서는 다음과 같다. 첫 번째로 ATP 비의존적 기작(ATP-independent mechanism)을 통한 단백질-DNA 복합체(protein-dna complex)의 형성이 손상부위의 인식(damage recognition)에 관여 한다. 두 번째로 ATP가수분해효소 일부(ATPase subunit)와의 결합을 통해 DNA 풀림(unwind), 풀린 이중나선 형태(open structure)를 형성을 한다. 세 번째로 3’ -> 5’ 의 순차적인 dual incision을 통해 손상을 제거한다. 아지즈 산자르의 nucleotide excision repair(NER) 기작은 DNA 분자 단위에서의 기본적인 복구과정을 규명했다. 손상부위의 인식(damage recognition)부터 절제 수선 과정(excision repair)까지의 전반적인 연구를 통해 세포 안에서 일어나는 DNA의 수선에 대한 기반을 제공한 것이다. 이로 인해 암 치료법 등 많은 DNA-level에서의 활용법의 가능성을 제시하였고 현재도 많은 화재가 되고 있다. 앞으로 연구가 활발히 될 분야이며 특히 인간에서의 기작은 아직 완전히 밝혀지지 않았기에 더욱 더 활발히 연구될 분야라고 볼 수 있다.
손상부위의 인식(Damage Recognition)
손상부위의 DNA는 정상적인 부위의 DNA와 다른 구조를 보인다. 그들은 구조적으로 뒤틀려있거나 튀어나와있기 때문에, 이와 결합할 수 있는 특이적 단백질이 이 구조들에 결합하면서 손상부위를 인식한다. 주로 zinc finger, helix turn helix가 DNA에 bind하는 주요 단백질의 motif가 된다.helix turn helix는 단백질의 한 부분으로써 일반적으로 DNA에 특이적으로 혹은 비특이적으로 결합하는 역할을 한다. 손상되는 부위의 인식은 이들의 특이성을 통해 일어나고, 그 인식을 정확하고, 빠르게 진행하기 위해 아래의 전략이 필요한 것이다.
절제 수선(Excision Repair)
본격적으로 손상 인식(damage recognition) 후에 절제 수선이 일어난다. 대장균과 인간에서 기구는 다르지만 비슷한 기작으로 repair가 일어난다. helicase로 DNA를 풀어내고 절개(incision) 시켜 문제가 있는 DNA 염기를 잘라낸다.
대장균에서의 절제 수선(Excision Repair in E.coli)

대장균에서의 절제 수선(Excision Repair in E.coli)
절제 수선(Excision repair)는 처음에 E.coli에서 발견되었고, UvrA, UvrB, UvrC에 의해 일어난다는 것이 밝혀졌다. 손상부위의 5’부근, 3’부근의 두 곳을 인식하여 12-13 뉴클레오티드(nucleotide)의 염기(bases)를 들어낸다. 이것을 ABC excision nuclease라고 부른다. 각각의 기능은 다음과 같다.
- UvrA : zinc finger, helix turn helix가 dna binding 을 하고, leucine zipper가 dimerization에 관여한다, ATP 존재하에 dimerization되고, 그후 UvrB와 함께 A2B1 complex를 형성한다. Molecular matchmaker의 기능을 한다.
- UvrB : SNF2 helicase family의 일종으로 제한적인 기능만 수행한다.(DNA unwinding, DNA-dependent ATPase) UvrB이 excision repair에서 핵심적인 기능을 수행하지만 혼자서는 아무것도 하지 못한다. B-hairpin motif가 DNA 결합(binding)에 역할을 한다.
- UvrC : UvrB와는 약하게 반응하지만 UvrB-DNA complex에는 강하게 반응한다. 2개의 helix-hairpin-helix motif가 c-terminal에 존재하고, DNA 결합(binding)과 dual incision에 중요하다.
- UvrD : SNF1 helicase
절제수선의 기작은 다음과 같다.
- 먼저 A2B1 복합체가 형성된 후에 DNA에 이 복합체가 결합한다.
- UvrB가 DNA의 이중나선을 풀면 UvrA 이합체가 분해되고 UvrC가 모집된다.
- 이후에는 dual incision이 일어나고 UvrD와 함께 잘려나간 염기를 제거한다.
- 최종적으로 Pol 1 ligase가 새로운 가닥을 만들면서 gap을 채워넣게 된다.
인간에서의 절제 수선(Excision Repair in Humans)

인간에서의 절제수선, global genomic repair과정을 보여준다 전사와 관련이 없이 작동할 수 있으며 XPC-Rad23B complexes에 의해 인식되면서 작동된다.
E coli와 달리 인간에서는 6개의 수선 요소(repair factor)들이 있으며 그 중 3개는 손상 인식(damage recognition), TFIIH는 helicase, 그리고 2개는 incision (절개)를 담당한다. 매커니즘은 다음과 같다
- XPA, RPA XPC-TFIIH가 랜덤한 순서로 목표 지역(lesion site)에 집합한다.
- XPA, RPA, XPC의 협동적 상호작용(cooperative interaction)과 TFHII의 kinetic proofreading activity(오류를 찾는 과정)에 의해 손상 부위(damage site)에 대한 특이성(specificity)를 형성한다.
- 그 다음 TFIIH가 ATP 분해로 DNA를 unwinding하여 bubble을 형성하는데 이 때의 complex를 PIC I이라 칭한다.
- 이 후 XPC가 XPG로 교체되어 PIC3 complex를 형성한다. 이 때 절개가 일어나 24-32nt의 oligomer가 나오고 생긴 빈공간(gap)은 RFC/PCNA, Pol delta-epsilon에 의해 매꿔진다. 마지막으로 DNA (ligase)가 봉합한다.
Transcription coupled repair

TC(transcription coupled) excision repair 전사와 연결되어 작동하는 절제 수선과정을 보여준다
excision repair은 사실 다른과정과 연결되어 있으며 상당부분 연결된 방식으로 일어난다. NER은 RNA 중합효소의 전사와 연결이 되어 있으며 전사로 인해 수선(repair)이 촉진된다. 그렇기 때문에 전사가 일어나는 전사된 가닥(transcribed strand)에서만 절제 수선(excision repair)이 일어날 수 있다.
대장균의 경우(in E.coli)
E. coli에서 절제 수선(Excision repair)과 전사는 transcription repair coupling factor (TRCF)로 인해 연결된다. TRCF는 RNA 중합효소와 UvrA 양쪽에 친화성(affinity)을 가져 서로를 연결 시킬 수 있다. 이 전체적인 과정에서 rate-limiting step은 damage recognition이며 오직 전사가닥(transcribing strand)에서만 수선(repair)가 일어난다.
DNA lesion이 비전사 가닥(nontranscribed strand)에 있을 때 전사(transcription)는 진행되지만 DNA lesion이 전사가닥(transcribed strand)에 있을 경우 RNA 중합효소는 손상 부위(damage site)에서 멈추게 되고 TRCF가 붙는다. TRCF는 RNA 중합효소와 완전히 조합되지 않은 전사체(transcript)를 떨어뜨리며 동시에 UVR A2B1 complex를 모집(recruiting) 한다. 이후 TRCF는 떨어지며 Uvr complex가 손상 부위(damage site)에 loading된다. TRCF가 떨어진 후 UvrB가 손상 부위(damage site)에 loading 되며 절제 수선(excision repair)가 시행 된다.
사람의 경우(in Humans)
거의 대부분 밝혀진 E. coli와 달리 인간에서 전사 짝지음(transcription coupling)은 아직 정확히 밝혀지지 않았다. 이에 노벨상 수상자인 아지즈 산자르는 두 가지 모델을 제시한다.
첫 번째 모델은 그림 왼쪽에 나타나듯이 수선 요소(repair factor)들이 멈춘 RNA 중합효소와 전사체(transcript)의 존재와 상관 없이 손상 부위(damage site)에 가서 조립 후 수선(repair)을 진행하는 모델이다. 즉 RNA 중합효소를 가닥(strand)에서 떼어내지 않고 수선(repair)하는 모델이며, 이 과정이 끝날 경우 중합효소(polymerase)가 멈췄던 자리에서 다시 전사를 재기한다. 이 모델에서는 CSA, CSB가 linker로서 작용하는 것으로 추정된다.
두 번째 모델은 그림 오른쪽에 나온 것이다. 여기서는 TTF2라는 요소가 중합효소(polymerase)를 떨어뜨린 후 수선 요소(repair factor)들이 와서 NER를 하는 모델입이다. TTF2는 E. coli에서의 TRCF와 동일한 역할을 담당한다. 수선(Repair)이 끝나면 전사는 처음부터 다시 시작된다.
참조
- Joyce T. Reardon, Aziz Sancar (2005). Nucleotide Excision Repair. Progress in Nucleic Acid Research and Molecular Biology 79, 183-235
- Aziz Sancar, Joyce T. Reardon (2004). Nucleotide Excision Repair in E. Coli and Man. Advances in Protein Chemistry 69, 43-71
- Laura A. Lindsey-Boltz, Michael G. Kemp, Jinchuan Hu, and Aziz Sancar (2015). Analysis of ribonucleotide removal from DNA by human nucleotide excision repair. J. Biol. Chem. published online October 21, 2015
| 위키미디어 공용에 관련된 미디어 분류가 있습니다. 아지즈 산자르 |
분류:
- 1946년 태어남
- 살아있는 사람
- 터키의 생화학자
- 터키의 의학자
- 터키의 무슬림
- 터키의 노벨상 수상자
- 노벨 화학상 수상자
- 이스탄불 대학교 동문
- 텍사스 대학교 동문
- 터키계 미국인
- 미국 과학 아카데미의 회원
- 미국 예술과학 아카데미 석학회원
- 이스탄불 공과대학교 동문
(RLQ=window.RLQ||[]).push(function()mw.config.set("wgPageParseReport":"limitreport":"cputime":"0.188","walltime":"0.269","ppvisitednodes":"value":442,"limit":1000000,"ppgeneratednodes":"value":0,"limit":1500000,"postexpandincludesize":"value":51624,"limit":2097152,"templateargumentsize":"value":1120,"limit":2097152,"expansiondepth":"value":9,"limit":40,"expensivefunctioncount":"value":0,"limit":500,"unstrip-depth":"value":0,"limit":20,"unstrip-size":"value":0,"limit":5000000,"entityaccesscount":"value":1,"limit":400,"timingprofile":["100.00% 149.439 1 -total"," 51.87% 77.509 1 틀:위키공용분류"," 46.79% 69.922 1 틀:Sister"," 44.61% 66.669 1 틀:사이드_박스"," 21.14% 31.585 1 틀:Llang"," 19.50% 29.142 2 틀:둘러보기_상자"," 16.77% 25.063 1 틀:노벨_화학상_수상자"," 9.82% 14.676 1 틀:2015년_노벨상_수상자"," 7.97% 11.903 1 틀:연도별_노벨상_수상자"," 6.71% 10.029 1 틀:Lang"],"scribunto":"limitreport-timeusage":"value":"0.049","limit":"10.000","limitreport-memusage":"value":1546023,"limit":52428800,"cachereport":"origin":"mw1332","timestamp":"20190709114700","ttl":86400,"transientcontent":true););"@context":"https://schema.org","@type":"Article","name":"uc544uc9c0uc988 uc0b0uc790ub974","url":"https://ko.wikipedia.org/wiki/%EC%95%84%EC%A7%80%EC%A6%88_%EC%82%B0%EC%9E%90%EB%A5%B4","sameAs":"http://www.wikidata.org/entity/Q15118973","mainEntity":"http://www.wikidata.org/entity/Q15118973","author":"@type":"Organization","name":"uc704ud0a4ubbf8ub514uc5b4 ud504ub85cuc81dud2b8 uae30uc5ecuc790","publisher":"@type":"Organization","name":"Wikimedia Foundation, Inc.","logo":"@type":"ImageObject","url":"https://www.wikimedia.org/static/images/wmf-hor-googpub.png","datePublished":"2015-10-11T04:57:53Z","dateModified":"2019-04-26T01:41:33Z"(RLQ=window.RLQ||[]).push(function()mw.config.set("wgBackendResponseTime":141,"wgHostname":"mw1330"););
